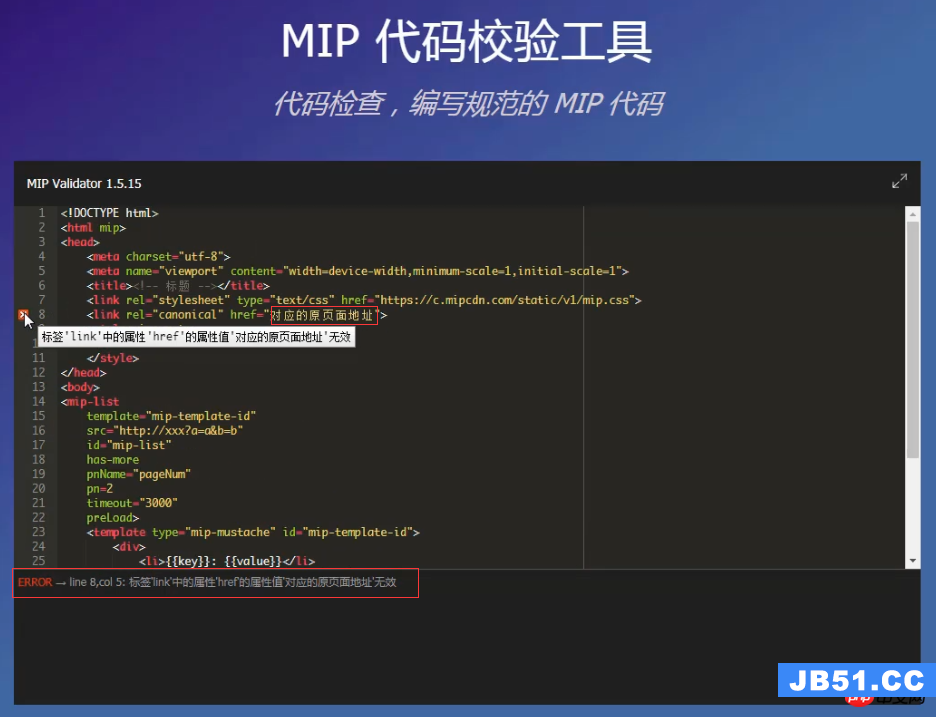
(我已经尝试过在
biostar上问过这个问题了,但是由于文本挖掘中的某个人认为有更好的解决方案的可能性很小,我也在这里重新发布)
我试图实现的任务是对齐几个序列.
我没有匹配的基本模式.我所知道的只是“真实”模式的长度应为“30”,并且我在随机点引入了缺失值的序列.
这是一个这样的序列的例子,在左边我们看到了缺失值的真实位置,在右边我们看到了我们能够观察到的序列.
我的目标是仅使用我在右栏中获得的序列重建左列(基于每个位置中的许多字母相同的事实)
Real_sequence The_sequence_we_see 1 CGCAATACTAAC-AGCTGACTTACGCACCG CGCAATACTAACAGCTGACTTACGCACCG 2 CGCAATACTAGC-AGGTGACTTCC-CT-CG CGCAATACTAGCAGGTGACTTCCCTCG 3 CGCAATGATCAC--GGTGGCTCCCGGTGCG CGCAATGATCACGGTGGCTCCCGGTGCG 4 CGCAATACTAACCA-CTAACT--CGCTGCG CGCAATACTAACCACTAACTCGCTGCG 5 CGCACGGGTAAGAACGTGA-TTACGCTCAG CGCACGGGTAAGAACGTGATTACGCTCAG 6 CGCTATACTAACAA-GTG-CTTAGGC-CTG CGCTATACTAACAAGTGCTTAGGCCTG 7 CCCA-C-CTAA-ACGGTGACTTACGCTCCG CCCACCTAAACGGTGACTTACGCTCCG
以下是重现上述示例的示例代码:
ATCG <- c("A","T","C","G")
set.seed(40)
original.seq <- sample(ATCG,30,T)
seqS <- matrix(original.seq,200,T)
change.letters <- function(x,number.of.changes = 15,letters.to.change.with = ATCG)
{
number.of.changes <- sample(seq_len(number.of.changes),1)
new.letters <- sample(letters.to.change.with,number.of.changes,T)
where.to.change.the.letters <- sample(seq_along(x),F)
x[where.to.change.the.letters] <- new.letters
return(x)
}
change.letters(original.seq)
insert.missing.values <- function(x) change.letters(x,3,"-")
insert.missing.values(original.seq)
seqS2 <- t(apply(seqS,1,change.letters))
seqS3 <- t(apply(seqS2,insert.missing.values))
seqS4 <- apply(seqS3,function(x) {paste(x,collapse = "")})
require(stringr)
# library(help=stringr)
all.seqS <- str_replace(seqS4,"-","")
# how do we allign this?
data.frame(Real_sequence = seqS4,The_sequence_we_see = all.seqS)
我明白,如果我所拥有的只是一个字符串和一个模式,我就可以使用
library(Biostrings) pairwiseAlignment(...)
但在我提出的情况下,我们正在处理许多序列以相互对齐(而不是将它们与一个模式对齐).
在R中有没有已知的方法?
谢谢,
塔尔
解决方法
虽然这是一个相当古老的线程,但我不想错过机会提及,因为Bioconductor 3.1,有一个包’msa’实现了三种不同的多序列比对算法的接口:ClustalW,ClustalOmega和MUSCLE.该软件包可在所有主要平台(Linux / Unix,Mac OS和Windows)上运行,并且在您无需安装任何外部软件的意义上是自包含的.更多信息可在
http://www.bioinf.jku.at/software/msa/和
http://www.bioconductor.org/packages/release/bioc/html/msa.html找到.
版权声明:本文内容由互联网用户自发贡献,该文观点与技术仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容, 请发送邮件至 dio@foxmail.com 举报,一经查实,本站将立刻删除。